Tách chiết DNA/RNA – 04 phương pháp tách chiết phổ biến nhất
Tách chiết thô (crude)
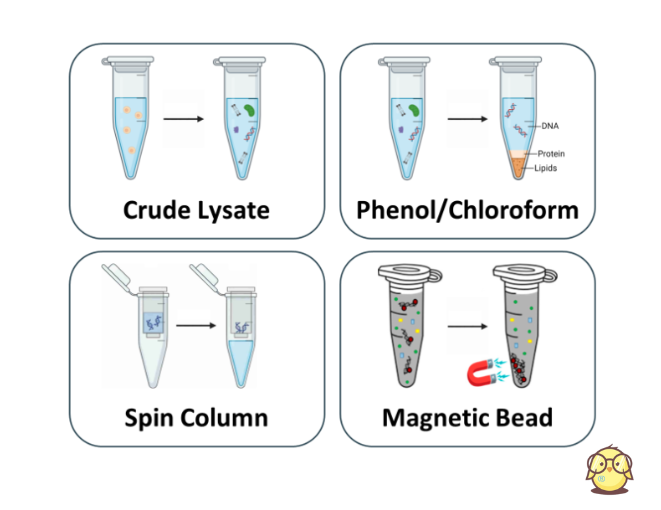
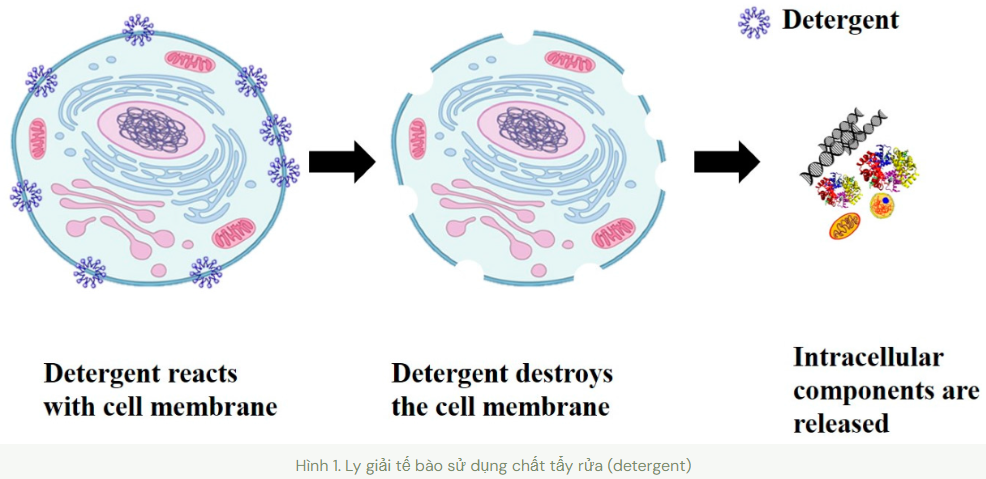
Một quy trình tách chiết DNA/RNA cơ bản sẽ bao gồm 3 bước: 1-ly giải tế bào, 2-loại bỏ các tạp chất và 3-cô đặc và thu hồi. Tuy nhiên, tách chiết thô chỉ có bước ly giải tế bào (Hình 1).
Ưu điểm
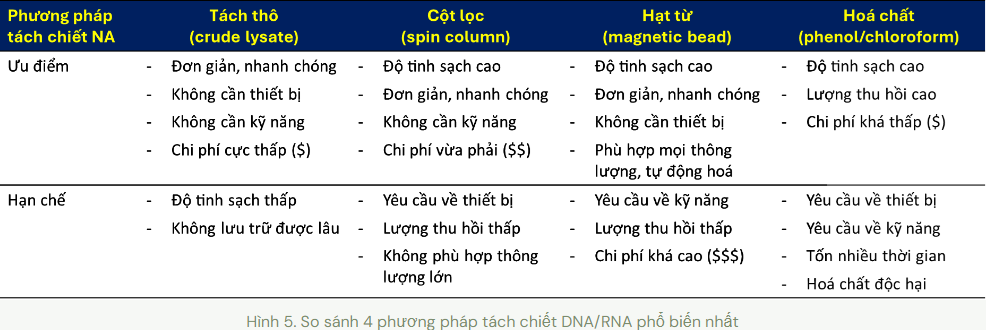
- Quy trình đơn giản, nhanh chóng
- Không yêu cầu về thiết bị
- Không đòi hỏi về kỹ năng thao tác
- Chi phí tách chiết DNA/RNA cực kỳ thấp
Hạn chế
- Độ tinh sạch thấp
- Không thể lưu trữ dài hạn
Bởi vì độ tinh sạch thấp, có khá ít ứng dụng phù hợp với tách chiết thô. Ứng dụng phù hợp có thể kế đến là các kỹ thuật khuếch đại nucleic acid như PCR, realtime PCR, LAMP, RPA, …
Tách chiết bằng cột lọc (spin column)
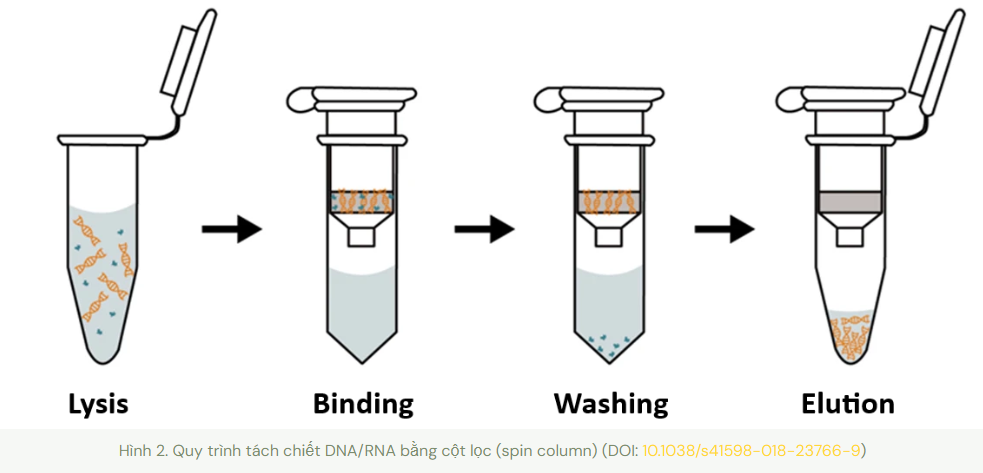
Phương pháp này sử dụng một cột lọc có một lớp màng có ái lực cao với nucleic acid để tách DNA/RNA ra khỏi các tạp chất khác trong mẫu sau khi đã ly giải tế bào. Chất liệu màng phổ biến nhất là silica, gần đây thì có thêm màng silicon carbide (SiC) chuyên dụng cho tách chiết RNA.
Quy trình tách chiết sử dụng cột lọc có thể tóm tắt bằng 4 từ tiếng Anh: Lysis-Binding-Washing-Elution. Trong đó bước 2-4 được thực hiện bằng cách chuyển mẫu/hoá chất lên cột lọc, sau đó ly tâm.
- Lysis – Ly giải tế bào
- Binding – Bắt giữ DNA/RNA trên cột lọc
- Washing – Rửa bỏ các tạp chất trên cột lọc
- Elution – Rửa giải DNA/RNA ra khỏi cột lọc
Ưu điểm
- Độ tinh sạch cao
- Quy trình đơn giản, nhanh chóng
- Không đòi hỏi về kỹ năng thao tác
- Chi phí tách chiết ở mức vừa phải
Hạn chế
- Yêu cầu về thiết bị (máy ly tâm)
- Không phù hợp thông lượng mẫu lớn
- Lượng DNA/RNA thu hồi khá thấp
Tách chiết bằng hạt từ (magnetic bead)
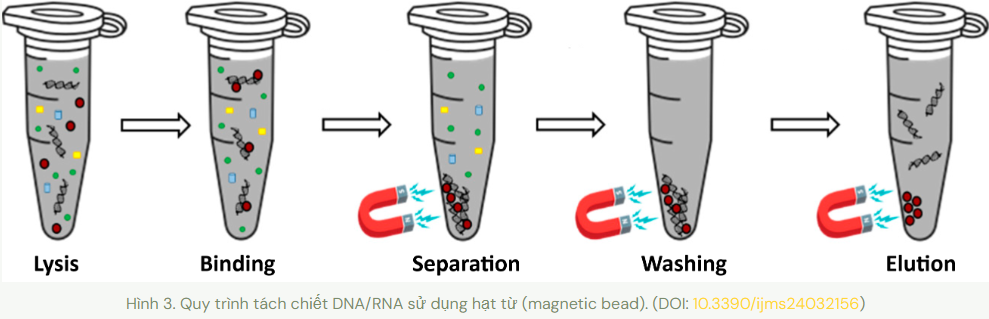
Phương pháp này sử dụng các hạt từ có ái lực cao với nucleic acid để tách DNA/RNA ra khỏi các tạp chất khác trong mẫu sau khi đã ly giải tế bào. Chất liệu bề mặt hạt từ thường là silica hoặc kháng thể bắt DNA.
Bởi vì không yêu cầu về thiết bị, quy trình tách chiết hạt từ phù hợp với mọi thông lượng mẫu và dễ dàng tự động hoá. Đó cũng là lý do khiến phương pháp này ngày càng được sử dụng rộng rãi.
Quy trình tách chiết sử dụng hạt từ có thể tóm tắt bằng 4 từ tiếng Anh: Lysis-Binding-Washing-Elution. Trong đó bước 2-4 được thực hiện bằng cách kết hợp các hạt từ, hoá chất và nam châm (Hình 3).
- Lysis – Ly giải tế bào
- Binding – Bắt giữ DNA/RNA trên hạt từ
- Washing – Rửa bỏ các tạp chất trên hạt từ
- Elution – Rửa giải DNA/RNA ra khỏi hạt từ
Ưu điểm
- Độ tinh sạch cao
- Quy trình đơn giản, nhanh chóng
- Không yêu cầu về thiết bị
- Phù hợp mọi thông lượng, dễ dàng tự động hoá
Hạn chế
- Chi phí tách chiết ở mức cao
- Lượng DNA/RNA thu hồi khá thấp
- Yêu cầu nắm được kỹ năng thao tác
Tách chiết bằng phenol/chloroform
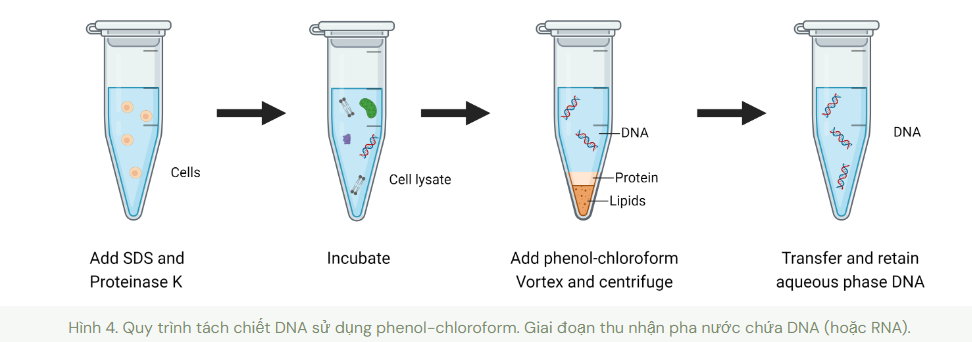
Phương pháp này sử dụng 3 loại hoá chất là phenol/chloroform/isoamyl alcohol (25:24:1) kết hợp với ly tâm để tách hỗn hợp sau ly giải thành 2 pha, được ngăn cách bởi lớp tủa protein mỏng (Hình 4).
- Pha nước ở phía trên chứa DNA (hoặc RNA)
- Pha hữu cơ ở phía dưới chứa phenol và lipid
Pha nước chứa DNA (hoặc RNA) sẽ được tinh sạch bằng cách kết tủa sử dụng ethanol (hoặc isopropanol) kết hợp với ly tâm. Cuối cùng, phần tủa DNA/RNA tinh sạch được thu hồi bằng cách tái hoà tan trong nước hoặc đệm rửa giải (elution buffer).
Ưu điểm
- Độ tinh sạch cao
- Lượng DNA/RNA thu hồi cao
- Chi phí tách chiết ở mức thấp
Hạn chế
- Yêu cầu về thiết bị (máy ly tâm)
- Tốn nhiều thời gian
- Hoá chất độc hại cho sức khoẻ
- Yêu cầu nắm được kỹ năng thao tác
Tổng kết các ưu điểm và hạn chế
Tài liệu tham khảo
Ali, N., Rampazzo, R.D.C.P., Costa, A.D.T. and Krieger, M.A., 2017. Current nucleic acid extraction methods and their implications to point‐of‐care diagnostics. BioMed research international, 2017(1), p.9306564.
Dhaliwal, A., 2013. DNA extraction and purification. Mater Methods, 3, p.191.
Li, P., Li, M., Yue, D. and Chen, H., 2022. Solid‐phase extraction methods for nucleic acid separation. A review. Journal of Separation Science, 45(1), pp.172-184.
Prins, T., Broothaerts, W., Burns, M., Demšar, T., Edelmann, S., Papazova, N., Peterseil, V. and Taverniers, I., 2024. Guidance on the selection and use of DNA extraction methods (No. KJ-NA-31-809-EN-N). Publications Office of the European Union.
Shehadul Islam, M., Aryasomayajula, A. and Selvaganapathy, P.R., 2017. A review on macroscale and microscale cell lysis methods. Micromachines, 8(3), p.83.
Ye, X. and Lei, B., 2023. The current status and trends of DNA extraction. Bioessays, 45(8), p.2200242.
Chia sẻ: